En una época caracterizada por la rápida convergencia de la tecnología y la ciencia, el término “Biohacking” ha surgido como un fenómeno fascinante y controvertido. Los Biohackers, personas que exploran la biología utilizando métodos y técnicas no convencionales, han comenzado a revolucionar nuestra comprensión de las ciencias de la vida. Junto a este movimiento, el desarrollo de herramientas y lenguajes de programación adaptados a la investigación biológica, como Biopython, ha facilitado un progreso sin precedentes en este campo. Profundicemos en el mundo de los Biohackers, examinemos sus motivaciones, métodos y contribuciones, al mismo tiempo que exploremos la importancia de Biopython en el avance de la investigación y la innovación biológicas.
El auge de los Biohackers
Los Biohackers, a menudo identificados como “científicos” que desdibujan los límites entre los investigadores tradicionales y los aficionados. Estas personas provienen de diversos orígenes, que incluyen informática, ingeniería, medicina y más. Su pasión compartida radica en aplicar técnicas no convencionales a la investigación biológica, a menudo aprovechando herramientas de código abierto y tecnologías fácilmente accesibles. Una de las fuerzas impulsoras detrás del auge de los Biohackers es la democratización del conocimiento y el acceso a los recursos, lo que permite a las personas involucrarse con conceptos científicos complejos fuera de las instituciones académicas tradicionales.
Las motivaciones para ingresar al mundo del Biohacking son diversas. Algunos están motivados por la curiosidad y buscan descubrir los misterios de la biología a través de la experimentación práctica. Otros se inspiran en el deseo de abordar desafíos globales apremiantes, como la degradación ambiental o problemas de salud pública. Aun así, otros ven el Biohacking como un medio de experimentación para hacer modificaciones genéticas personalizadas para mejorar las capacidades de los organismos vivos. Si bien estas motivaciones varían ampliamente, colectivamente contribuyen a un panorama dinámico y de rápida evolución en las ciencias de la vida.
Métodos y controversias
Los Biohackers emplean una variedad de métodos no convencionales, a menudo utilizando equipos de laboratorio adquiridos a través de crowdfunding, colaboraciones o fabricación de bricolaje. Algunos de estos métodos incluyen la ingeniería genética, la biología sintética y la manipulación de microorganismos. Por ejemplo, los Biohackers han creado con éxito plantas bioluminiscentes, organismos modificados para producir compuestos novedosos e incluso bacterias modificadas para detectar contaminantes ambientales.
Sin embargo, la naturaleza poco convencional del Biohacking ha generado preocupaciones éticas y de seguridad. El potencial de consecuencias no deseadas, como la liberación de organismos genéticamente modificados en el medio ambiente, plantea dudas sobre la supervisión regulatoria y la realización responsable de los experimentos de Biohacking. Como resultado, las discusiones en torno a la bioética y la regulación de las actividades de Biohacking se han vuelto esenciales para garantizar el impacto positivo de estos esfuerzos.
Biopython: empoderando a Biohackers e investigadores
Junto con el auge de los Biohackers, el campo de la biología computacional ha experimentado un crecimiento notable. Biopython, un lenguaje de programación especializado, ha surgido como piedra angular en este dominio. Biopython es un proyecto de código abierto que proporciona un conjunto integral de herramientas, bibliotecas y módulos para aplicaciones de bioinformática y biología computacional. Simplifica el proceso de análisis de datos biológicos, realización de simulaciones y automatización de tareas, lo que lo convierte en un recurso invaluable para investigadores, Biohackers y biólogos tradicionales por igual.
La versatilidad de Biopython es evidente en su amplia gama de funcionalidades. Admite tareas como el análisis de secuencias, la predicción de estructuras, la visualización de datos e incluso la simulación de sistemas biológicos. Su sintaxis fácil de usar, su extensa documentación y el apoyo activo de la comunidad reducen la barrera de entrada, lo que permite a las personas sin experiencia formal en programación aprovechar su poder para la investigación biológica.
La sinergia revelada
La sinergia entre los Biohackers y Biopython destaca el potencial transformador de combinar la experimentación no convencional con herramientas informáticas avanzadas. Los Biohackers, equipados con habilidades prácticas y una profunda curiosidad por la biología, pueden aprovechar Biopython para procesar y analizar los datos generados a partir de sus experimentos. Esta integración permite una iteración rápida, pruebas de hipótesis y conocimientos basados en datos que las canalizaciones de investigación tradicionales podrían tener dificultades para lograr.
Por ejemplo, un Biohacker interesado en comprender la función de un gen específico podría emplear Biopython para analizar secuencias de ADN, predecir estructuras de proteínas y simular las interacciones del producto génico con otras moléculas. Tal enfoque acelera el proceso de descubrimiento y permite que los Biohackers contribuyan significativamente a nuestra comprensión de los sistemas biológicos.
Desafíos y direcciones futuras
Si bien la sinergia de los Biohackers y Biopython es una gran promesa, no está exenta de desafíos. La accesibilidad de las herramientas computacionales y de Biohacking requiere un marco sólido para las consideraciones éticas y la supervisión regulatoria. Las pautas y estándares claros son cruciales para garantizar la realización responsable de los experimentos y prevenir riesgos potenciales.
Además, a medida que el campo evoluciona, la integración de tecnologías más avanzadas, como el aprendizaje automático y la inteligencia artificial, podría mejorar aún más las capacidades de los Biohackers y los biólogos computacionales. Estas tecnologías podrían permitir predicciones más precisas, conocimientos más profundos a partir de conjuntos de datos complejos y la identificación de patrones novedosos en la información biológica.
La interacción dinámica entre los Biohackers y Biopython ejemplifica el poder transformador de la colaboración interdisciplinaria en la intersección de la biología y la programación. Los Biohackers, impulsados por la curiosidad y el ingenio, tienen el potencial de desbloquear nuevos conocimientos sobre biología a través de la experimentación no convencional. Al mismo tiempo, Biopython empodera tanto a los investigadores como a los Biohackers al proporcionar un conjunto de herramientas versátil para el análisis y la simulación computacional. A medida que esta sinergia continúa evolucionando, es esencial equilibrar la innovación con una conducta responsable para garantizar un impacto positivo tanto en el descubrimiento científico como en la sociedad en su conjunto. El futuro depara interesantes posibilidades para la fusión de la exploración no convencional y la destreza computacional en el ámbito de las ciencias de la vida.
Introducción a Biopython: un tutorial de análisis de secuencias de ADN
Biopython es una poderosa biblioteca de código abierto diseñada para facilitar tareas de biología computacional y bioinformática. Proporciona una amplia gama de herramientas, módulos y funcionalidades que simplifican el análisis de secuencias de ADN, la predicción de estructuras de proteínas, la manipulación de datos y más. En este tutorial, nos centraremos en los conceptos básicos de Biopython demostrando cómo leer una secuencia de ADN de un archivo, calcular su contenido de GC y generar su complemento inverso.
Biopython Tutorial
Instalación de Biopython
Antes de comenzar, asegúrese de tener instalado Biopython. Puedes instalarlo usando pip:

Trabajar con secuencias de ADN usando Biopython
Comenzaremos importando los módulos y funciones necesarios desde Biopython:

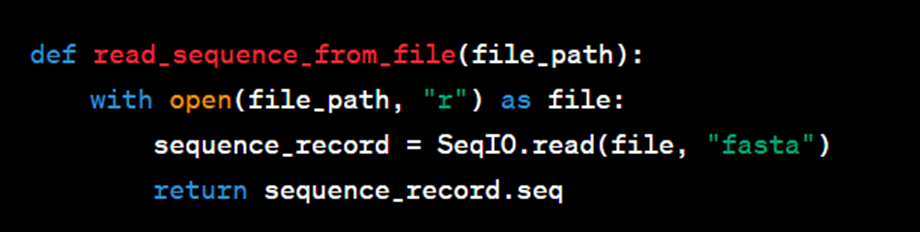
Lectura de la secuencia de ADN de un archivo
Crearemos una función que lea una secuencia de ADN de un archivo FASTA. Reemplace “wuhan.fasta” descargar aquí con la ruta a su archivo FASTA de entrada:
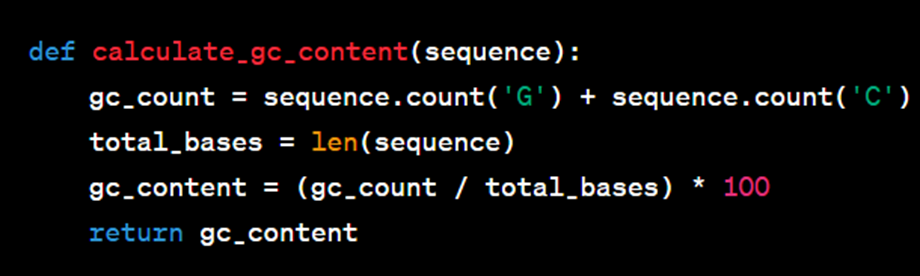
Cálculo del contenido de GC A continuación, calcularemos el contenido de GC de la secuencia de ADN. El contenido de GC es el porcentaje de bases que son guanina (G) o citosina (C). Crearemos una función para calcular
Generación de complemento inverso
El complemento inverso de una secuencia de ADN se obtiene invirtiendo la secuencia y reemplazando cada base con su base complementaria (A con T, C con G, G con C y T con A). Definiremos una función para generar el complemento inverso:

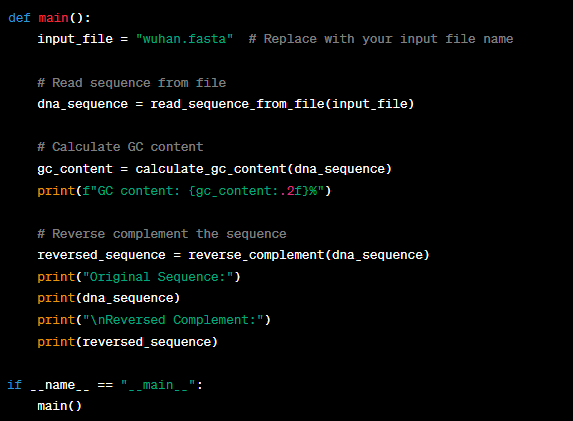
Poniéndolo todo junto – Función principal
Ahora, creemos una función principal que une todo. Reemplace “wuhan.fasta” con la ruta a su archivo FASTA de entrada:
Ejecutar el script
Guarde el script con una extensión .py (por ejemplo, wuhan.py) y ejecútelo usando su terminal o símbolo del sistema:

Respuesta:
‘Contenido de GC: 37,97 %’
“Contenido de GC: 37,97 %” es el resultado del cálculo realizado en la función compute_gc_content() del script.
Explicación:
El contenido de GC (contenido de guanina-citosina) de una secuencia de ADN se refiere al porcentaje de bases en la secuencia que son guanina (G) o citosina (C). En el ADN, G forma un par de bases con C y A forma un par de bases con T (timina).
En el script dado, la función compute_gc_content() calcula el contenido de GC contando las ocurrencias de bases G y C en la secuencia de ADN y luego dividiendo esa cuenta por el número total de bases en la secuencia. El resultado se multiplica por 100 para obtener el porcentaje.
La fórmula para calcular el contenido de GC es:
Contenido de GC (%) = (Número de bases G + Número de bases C) / Número total de bases * 100
Cuando ejecuta el script y proporciona una secuencia de ADN de un archivo (en este caso, “wuhan.fasta”), el script lee la secuencia, cuenta las ocurrencias de las bases G y C, calcula el contenido de GC usando la fórmula y luego imprime el resultado.
En el ejemplo dado, el contenido de GC de la secuencia de ADN en el archivo “wuhan.fasta” se calcula en aproximadamente 37,97 %. Esto significa que alrededor del 37,97 % de las bases de la secuencia son guanina (G) o citosina (C).
El especificador de formato .2f en la instrucción print() se usa para mostrar el resultado con dos decimales para una mejor legibilidad.
El contenido de GC del coronavirus 2 del Síndrome Respiratorio Agudo Severo (SARS-CoV-2) aísla Wuhan-Hu-1, el genoma completo que tiene un contenido de GC de aproximadamente el 37,97% tiene ciertas implicaciones biológicas y proporciona información sobre las características del material genético del virus. .
1. Rango típico: el contenido de GC del 37,97 % para el SARS-CoV-2 es relativamente moderado y se encuentra dentro del rango observado para muchos virus. Los virus pueden exhibir una amplia gama de porcentajes de contenido de GC, y el contenido de GC del SARS-CoV-2 no es excepcionalmente alto o bajo.
2. Evolución viral: el contenido de GC puede verse influenciado por procesos evolutivos. Puede reflejar la adaptación del virus a su huésped, así como las presiones selectivas que ha experimentado con el tiempo. Un contenido de GC relativamente estable puede sugerir que el virus ha ido evolucionando gradualmente y manteniendo su estabilidad genética.
3. Estructura y función del genoma: el contenido de GC puede afectar la estructura y la función del genoma viral. Un contenido moderado de GC puede influir en la estabilidad del genoma viral, afectando su capacidad para replicarse con precisión. Además, el contenido de GC puede tener implicaciones sobre cómo se transcribe y traduce el virus en proteínas.
4. Análisis comparativo: para sacar conclusiones más significativas, es esencial comparar el contenido de GC del SARS-CoV-2 con otros virus o coronavirus relacionados. Esto puede ayudar a los investigadores a comprender si el contenido de GC se conserva en diferentes cepas y aislamientos o si existen variaciones que podrían ser biológicamente significativas.
5. Implicaciones diagnósticas y terapéuticas: si bien el contenido de GC por sí solo puede no proporcionar información diagnóstica o terapéutica definitiva, puede contribuir a nuestra comprensión de la biología del virus. Los investigadores podrían explorar cómo regiones específicas con contenido variable de GC contribuyen a la patogenicidad del virus, las interacciones con el huésped o las respuestas inmunitarias.
En el contexto del SARS-CoV-2, un contenido de GC de alrededor del 37,97 % es solo un aspecto de la composición genética del virus. Es parte del panorama más amplio que los investigadores consideran al estudiar la evolución, la estructura, la función y las posibles interacciones del virus con el huésped. La interpretación de este contenido de GC debe realizarse junto con otra información genética, molecular y epidemiológica para obtener una comprensión integral del SARS-CoV-2 y su comportamiento.
Referencias
Severe acute respiratory syndrome coronavirus 2 isolate Wuhan-Hu-1, complete genome
FASTA Format for Nucleotide Sequences

